Iros Barozzi, Ass.-Prof. PhD
Gruppenleiter
T: +43 (0)1 40160-57517
iros.barozzi@meduniwien.ac.at
ORCID: 0000-0003-0690-3473
Forschungsschwerpunkt
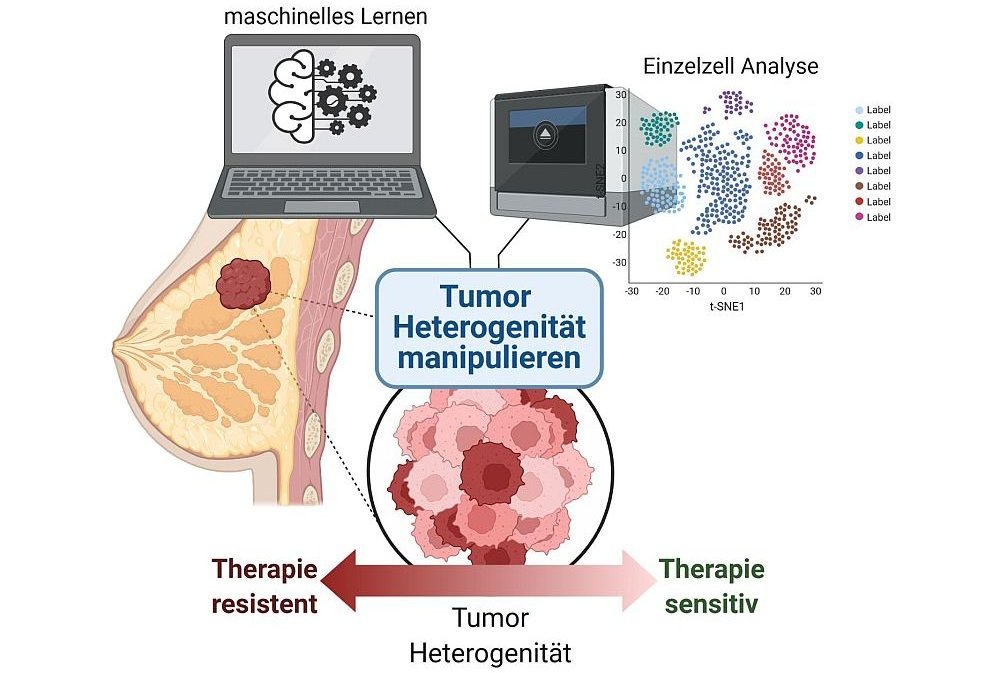
Ziel des Computational Cancer Genomics Laboratory ist es zu erforschen, inwiefern genregulatorische Netzwerke beim Fortschreiten von Brust- und Prostatakrebs zu arzneimittelresistenten, metastasierenden Erkrankungen fehlreguliert werden.
Ungeachtet der Schwierigkeiten bei der Identifizierung von Genen, die die Metastasenbildung vorantreiben, wurde dem Beitrag der transkriptionellen Heterogenität zur Resistenz im Vergleich zur genetischen Heterogenität bisher wenig Aufmerksamkeit geschenkt.
Wir glauben, dass wir durch die Verlagerung dieses Fokus neue Zusammenhänge zwischen der Heterogenität des Primärtumors und der Therapieresistenz identifizieren und nutzen könnten (Hong et al. 2019). Zu diesem Zweck integrieren wir Informationen sowohl auf Einzelzell- als auch auf Populationsebene durch Genomanalysen und verwenden maschinelles Lernen zur Vorhersage von Resistenzen.
Diese Ziele unterteilen die übergreifenden Themen der Arbeitsgruppe, nämlich die Identifizierung und Charakterisierung von Mustern transkriptioneller Heterogenität in scheinbar identischen Zellen und die Charakterisierung der zugrunde liegenden Regulationsmechanismen.
Wir glauben, dass ein besseres Verständnis dieser grundlegenden Mechanismen eine neue Perspektive bieten wird, um die Behandlung von primären Tumoren erfolgreich anzupassen und dadurch Resistenzen gegen aktuelle Therapien vorzubeugen.
Weiters nutzen wir unser Interesse für automatisierte Vorhersagen:
- der Funktion von nicht-kodierender, regulatorischer DNA (Barozzi et al. 2014, Mancino et al. 2015, Monti et al. 2017, Comoglio et al. 2019) und
- der Zielgene von Enhancern (Gorkin et al. 2020, Osterwalder et al. 2018).
Ausgewählte Publikationen
A functional survey of the regulatory landscape of estrogen-receptor-positive breast cancer evolution
Barozzi I#, Slaven N, Canale E, Lopes R, Monteiro Barbosa IA, Bleu M, Ivanoiu D, Pacini C, Mensa E, Chambers A, Bravaccini S, Ravaioli S, Gyorffy B, Dieci MV, Pruneri G, Galli GG#, Magnani L#
Cancer Discovery. 2024 May 16.
https://doi.org/10.1158/2159-8290.cd-23-1157
An atlas of dynamic chromatin landscapes in mouse fetal development
Gorkin DU*, Barozzi I*, Zhao Y*, Zhang Y*, Huang H*, Lee AY, Li B, Chiou J, Wildberg A, Ding B, Zhang B, Wang M, Strattan JS, Davidson JM, Qiu Y, Afzal V, Akiyama JA, Plajzer-Frick I, Novak CS, Kato M, Garvin TH, Pham QT, Harrington AN, Mannion BJ, Lee EA, Fukuda-Yuzawa Y, He Y, Preissl S, Chee S, Han JY, Williams BA, Trout D, Amrhein H, Yang H, Cherry JM, Wang W, Gaulton K, Ecker JR, Shen Y, Dickel DE, Visel A#, Pennacchio LA#, Ren B#.
Nature. 2020 Jul;583(7818):744-751.
http://doi.org/10.1038/s41586-020-2093-3
Single-cell transcriptomics reveals multi-step adaptations to endocrine therapy
Hong SP#, Chan TE, Lombardo Y, Corleone G, Rotmensz N, Bravaccini S, Rocca A, Pruneri G, McEwen KR, Coombes RC, Barozzi I#, Magnani L#.
Nature Communications. 2019 Sep 2;10(1):3840.
http://doi.org/10.1038/s41467-019-11721-9
Dissection of acute stimulus-inducible nucleosome remodeling in mammalian cells
Comoglio F*, Simonatto M*, Polletti S, Liu X, Smale ST, Barozzi I#, Natoli G#.
Genes & Development. 2019 Sep 1;33(17-18):1159-1174.
http://doi.org/10.1101/gad.326348.119
Enhancer redundancy provides phenotypic robustness in mammalian development
Osterwalder M, Barozzi I, Tissières V, Fukuda-Yuzawa Y, Mannion BJ, Afzal SY, Lee EA, Zhu Y, Plajzer- Frick I, Pickle CS, Kato M, Garvin TH, Pham QT, Harrington AN, Akiyama JA, Afzal V, Lopez-Rios J, Dickel DE#, Visel A#, Pennacchio LA#.
Nature. 2018 Feb 8;554(7691):239-243.
http://doi.org/10.1038/nature25461
Limb-Enhancer Genie: An accessible resource of accurate enhancer predictions in the developing limb
Monti R*, Barozzi I*#, Osterwalder M, Lee E, Kato M, Garvin TH, Plajzer- Frick I, Pickle CS, Akiyama JA, Afzal V, Beerenwinkel N, Dickel DE, Visel A#, Pennacchio LA#.
PLoS Computational Biology. 2017 Aug 21;13(8):e1005720.
http://doi.org/10.1371/journal.pcbi.1005720
Coregulation of transcription factor binding and nucleosome occupancy through DNA features of mammalian enhancers
Barozzi I*, Simonatto M*, Bonifacio S, Yang L, Rohs R, Ghisletti S, Natoli G.
Molecular Cell. 2014 Jun 5;54(5):844-857.
http://doi.org/10.1016/j.molcel.2014.04.006
(* co-first author; # co-corresponding author)