Optimierte Vorhersage von Cis-regulatorischen Elementen im Herzen
Wir erstellen einen Katalog von genregulatorischen Elementen in der menschlichen Herzentwicklung mit Einzelzellauflösung, indem wir Multi-omics-Daten mithilfe von maschinellem Lernen analysieren.
Weiters implementieren wir Modelle zur Vorhersage der Auswirkungen genetischer Variationen auf die Aktivität dieser Elemente. Das Projekt ist Teil des HeartX-Projekts (finanziert vom Schweizerischen Nationalfonds), einer Zusammenarbeit mit den Labors von Marco Osterwalder (https://www.osterwalderlab.com/) und Christian Zuppinger am Department for BioMedical Research (DBMR) der Universität Bern.
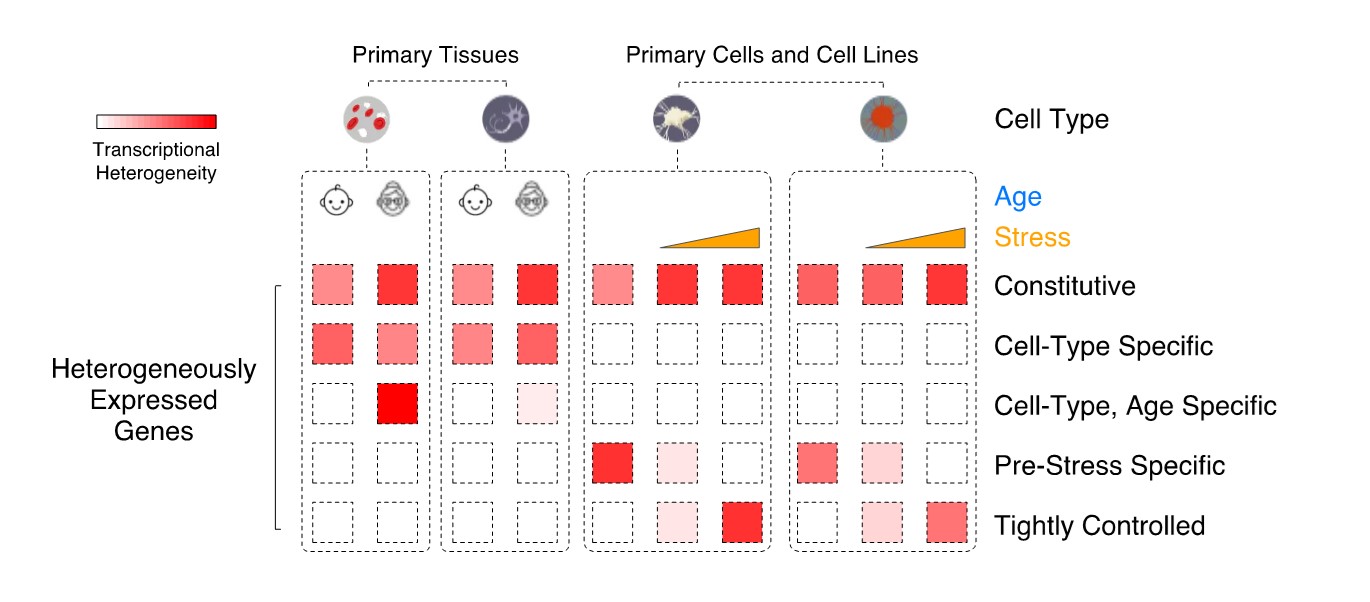
Regulierte Variabilität der Genexpression
Durch die Nutzung von Einzelzell-Transkriptionsdaten klassifizieren wir Säugetiergene nach dem jeweiligen Muster der transkriptionellen Heterogenität in Zellen des gleichen Typs, aus gesundem und krankem Gewebe (Abbildung 1). Das Ziel ist festzustellen, ob Unterschiede in der Variabilität durch verschiedene Regulationsmechanismen entstehen.
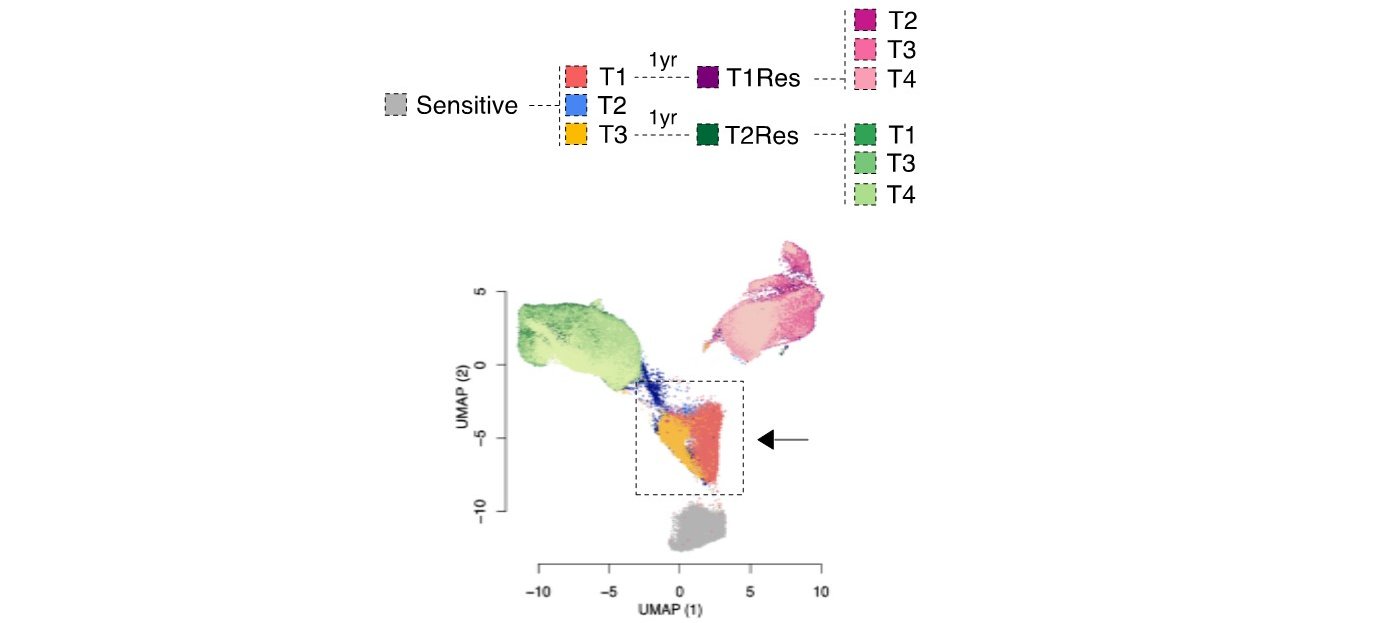
Ein konservierter Überlebensmechanismus von arzneimitteltoleranten Persisterzellen
Wir haben bereits eine Transkriptionssignatur identifiziert, die den Phänotyp dieser luminalen Brustkrebszellen vorhersagt, die kurzfristige endokrine Therapien überleben (Hong et al. 2019). Diese Zellen, die oft als arzneimitteltolerante Persisterzellen bezeichnet werden, führen letztlich zu einem Rückfall.
Unser Ziel ist:
- herauszufinden wie konserviert diese Signatur bei Tumoren unterschiedlichen Ursprungs ist, sowie ihre Bedeutung für das Überleben bei verschiedenen therapeutischen Strategien zu verstehen, und
- die Charakterisierung ihrer vorgeschalteten Regulatoren.
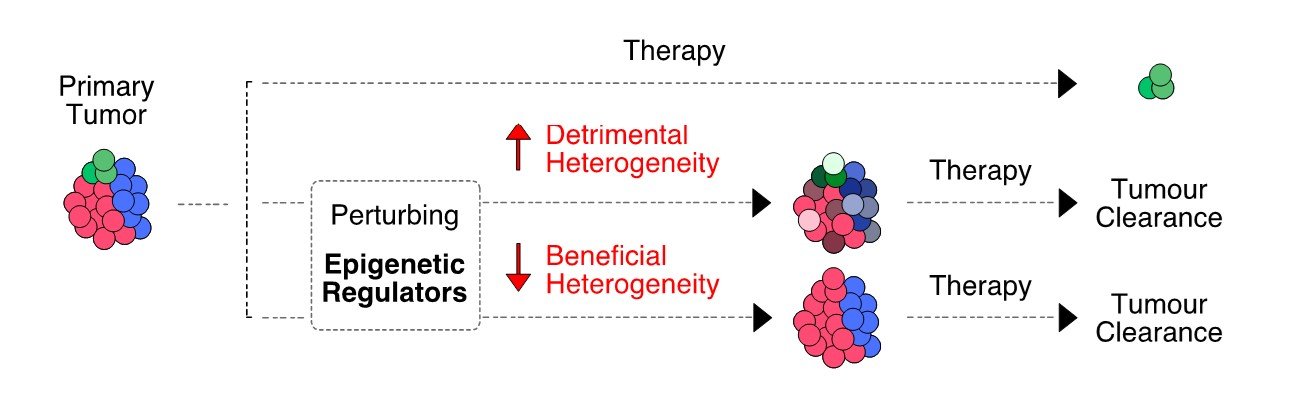
Manipulation der phänotypischen Heterogenität zur Verzögerung der Tumorprogression
Die Gene der sogenannten epigenetischen Regulatoren sind in Krebszellen oft mutiert oder fehlreguliert. Wir vermuten, dass ihre abweichende Aktivität mit transkriptionellen Fluktuationen in Zusammenhang steht, die zum Vorteil der Patient:innen manipuliert werden könnten (Abbildung 3). Basierend auf diesem Prinzip kombinieren wir In-vitro-Störungen (gerichtet auf epigenetische Regulatoren) mit Einzelzell-Transkriptomik, um neue Behandlungsstrategien zur Verzögerung der Tumorprogression zu entwickeln.
Entwicklung neuer Methoden zur Identifizierung der vorgeschalteten Regulatoren der Genaktivität
Wir nutzen zuvor veröffentlichte, validierte regulatorische Interaktionen, um maschinelle Lernmodelle zu trainieren, die dann neue, gewebespezifische Interaktionen im gesamten Genom vorhersagen.
Zu diesem Zweck kombinieren wir Co-Expressionsnetzwerke, Transkriptionsfaktor-Bindungsprofile und lokale Chromatin-Konformationsmessungen.